
DNA-sekvensering er i dag både et stort forskningsfelt og en viktig teknikk som er blitt uunnværlig for arkeologer, historikere, adferdsforskere, evolusjonsbiologer, cellebiologer, kreftforskere og -behandlere, rettsmedisinere, ikke minst hele livsvitenskap-feltet og så videre.
Kartlegging av organismers DNA har for eksempel vært brukt til å avsløre hvordan pesten og Svartedauden kom til Europa flere ganger under middelalderen. Forskere på UiOs Institutt for biovitenskap (IBV) har lagt fram DNA-bevis for at mektige vikinger på Island ble begravd sammen med hingster eller vallaker og at handel med hvalross-elfenben holdt liv i den norrøne bosettingen på Grønland.
IBV-forskerne har også produsert referansegenomer fra både Tore Torsk, Harald Harr og Lasse Laks og dokumentert de genetiske merkverdighetene i en liten tropisk fisk som aldri blir voksen. Et referansegenom er resultatet av en spesielt grundig analyse av genene hos et bestemt individ av en art, som for eksempel Tore Torsk, og senere kan referansen brukes til å sammenlikne med andre individer av samme art.
Men – hvordan foregår egentlig denne DNA-sekvenseringen? Hos mennesker består «livets kode» av til sammen over tre milliarder «bokstaver» fordelt på rundt 30 000 gener – som igjen er fordelt på 23 kromosomer. Det sier seg selv at det er krevende å skulle kartlegge rekkefølgen av mange milliarder bittesmå "bokstaver" i "livets kode" for hver enkelt art.
Enkel grunnkode, kompliserte byggverk
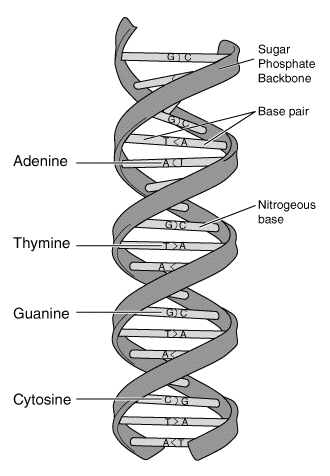
Det hele begynte for snart 66 år siden, da de britiske forskerne James Watson og Francis Crick oppdaget DNA-molekylets tredimensjonale struktur. Den 25. april 1953 publiserte de en artikkel på bare to sider (!) i tidsskriftet Nature, under den korte og fyndige tittelen “Molecular structure of Nucleic Acids”. Der beskrev de hvordan DNA-molekylet består av to lange tråder som er tvinnet rundt hverandre - og tvinnes fra hverandre når arvestoffet skal kopieres.
Det mest oppsiktsvekkende var kanskje at den genetiske koden – «livets kode» – på grunn-nivået er forbausende enkel og består av kun fire ulike basemolekyler, også kalt nukleotider. Disse fire molekylene utgjør bokstavene som livets kode er skrevet med, på et «ark» som består av de tvinnede trådene Watson og Crick oppdaget.
De fire molekyl-bokstavene heter adenin (A), cytosin (C), guanin (G) og tymin (T). Tre bokstaver ved siden av hverandre på DNA-tråden kalles et kodon, og hvert kodon fungerer som et kodeord for en bestemt aminosyre som skal brukes i proteinsyntesen. Det finnes bare 20 aminosyrer i menneskekroppen, men disse kan kombineres på så mange måter at det finnes til sammen rundt én million forskjellige proteiner.
Hvis kodonet for eksempel består av de tre base-molekylene guanin, cytosin og adenin i den rekkefølgen, betyr det at «proteinfabrikken» i cellen skal hente fram et molekyl av aminosyren alanin og bruke den som byggestein.
Det gikk lang tid etter 1953 før forskerne fant ut hvordan de skulle kartlegge rekkefølgen av A, C, G og T på de lange DNA-trådene. Det offentlig finansierte Human Genome-prosjektet (HUGO) startet formelt i 1990 med et budsjett på tre milliarder dollar og en tidsramme på 15 år. Prosjektet sorterte under det amerikanske National Institutes of Health og hadde deltakere fra USA, Kina, Frankrike, Japan og Storbritannia. Den første og ufullstendige «utskriften» av menneskets DNA-kode forelå i 2000.
Craig Venter fikk fart på sakene
– Men det skulle enda gå tre år før det siste humane kromosomet var ferdig analysert. Og det ville tatt enda lenger tid hvis ikke biokjemikeren og forretningsmannen Craig Venter hadde tatt opp konkurransen og utviklet en ny metode for DNA-kartlegging. Det er i prinsippet hans teknikk vi bruker i dag, forteller professor Kjetill S. Jakobsen ved CEES.
Kjetill S. Jakobsen er i dag en av de norske forskerne som har mest erfaring med DNA-kartlegging. Omkring 2005 begynte han å tenke på at kartlegging av arvestoffet hos viktige arter ville bli et stort og viktig forskningsfelt, og i 2007 gikk han til professor Nils Chr. Stenseth med en tung argumentasjon. Stenseth hadde nettopp fått en bevilgning fra Norges forskningsråd til det nye SFF-senteret CEES og fattet poenget med en gang.
Tospannet Nils Chr. Stenseth og Kjetill S. Jakobsen klarte deretter, med mye strev, å finansiere innkjøpet av en sekvenseringsmaskin – og så satte de i gang. I 2011 kom den første vitenskapelige artikkelen fra CEES-miljøet på trykk i Nature, med nyheten om at torsken har et helt spesielt immunsystem. Og deretter har resultatene fra DNA-kartleggerne ved CEES og Institutt for biovitenskap kommet som perler på en snor – eller som nukleotider på en DNA-streng.
Er du interessert i forskningsnyheter om realfag og teknologi: Følg Titan.uio.no på Facebook eller abonner på nyhetsbrevet vårt
Begynner med en liten kroppsdel
DNA-sekvenseringen begynner med at forskerne klipper ut en liten kroppsdel av en representant for den arten som skal undersøkes. Noen av disse representantene er etterhvert blitt kjendiser på sitt felt, som Tore Torsk og Harald Harr. Kroppsdelen kan deretter legges på sprit, som konserverer DNA-molekylene inntil de skal analyseres.
– Hvilken kroppsdel vi bruker, kan variere fra gang til gang. DNA-sekvensene er jo i prinsippet like i alle cellene i kroppen. Vi har brukt blod, blodrike organer, muskelvev og så videre. Da vi for eksempel skulle analysere genomet hos Tore Torsk, brukte vi DNA fra blodet hans. Men da vi skulle analysere andre torsker etterpå, klippet vi av en liten bit av en finne. Da hadde vi jo allerede et referansegenom fra Tore Torsk, slik at vi kunne stille mindre strenge krav til DNA-kvaliteten, forteller Jakobsen.
Det neste skrittet er å ekstrahere DNA fra kroppsvevet i prøven. Dette er en forholdsvis enkel prosess som til og med kan gjøres på et skolelaboratorium. Men deretter begynner det virkelig krevende arbeidet.
Kuttet alt opp i småbiter
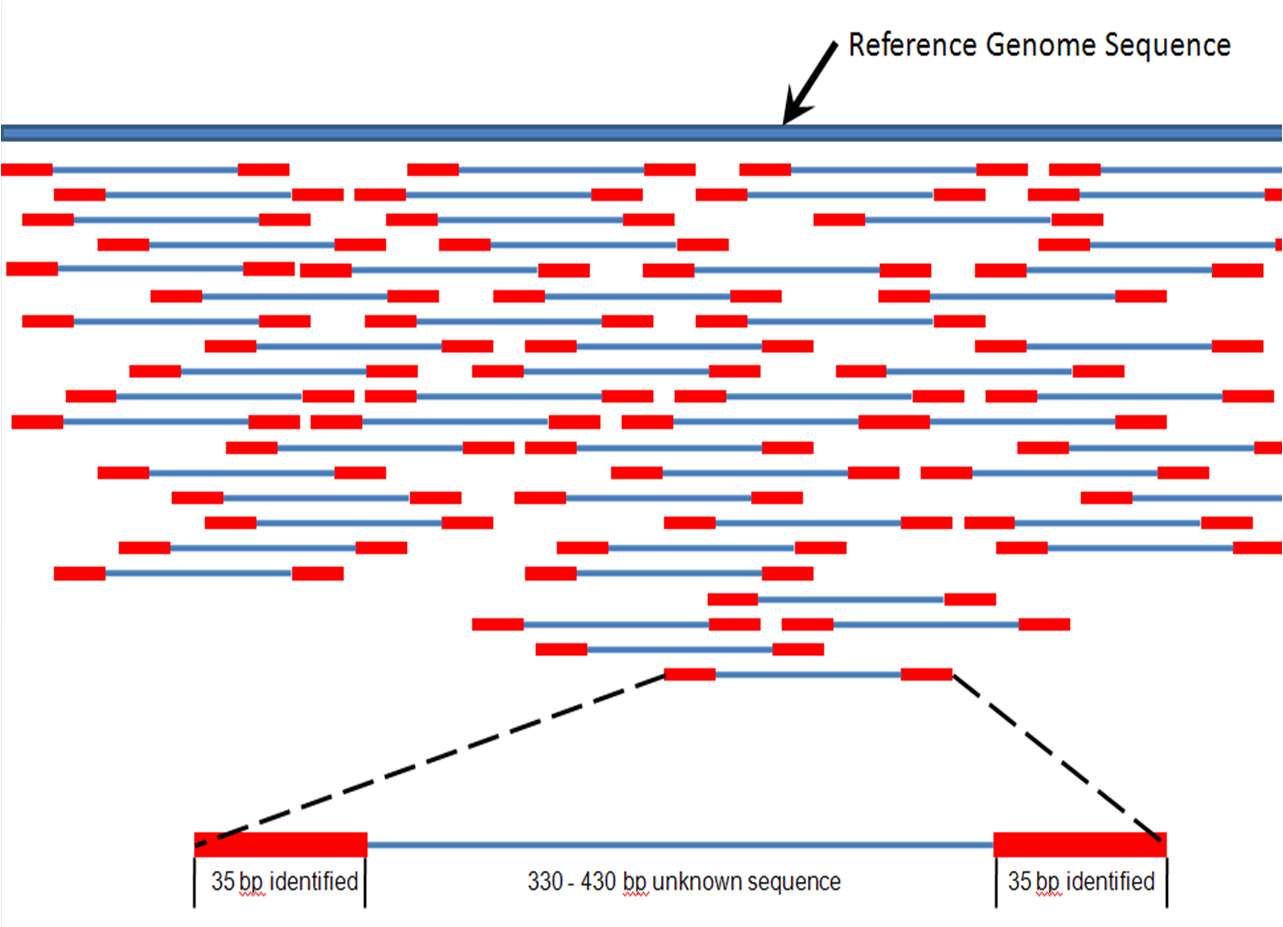
– Forskerne bak HUGO så på prosjektet som et månelandingsprogram og benyttet seg av en teknologi som i prinsippet «spaserte» langs DNA-strengene, kromosom for kromosom, og identifiserte ett nukleotid av gangen. Men det Craig Venter gjorde, var at han kuttet hele genomet opp i småbiter og sekvenserte hver enkelt småbit blindt, uten egentlig å vite hvor hver bit hørte hjemme. Teknikken kalles for Whole genome shotgun sequencing, forteller Jakobsen.
Grunnen til at Craig Venters hagle-tilnærming er bedre enn HUGO-tilnærmingen, er at det finnes grenser for hvor lange «bokstaver» som kan leses sammenhengende.
– På den tiden kunne man bare lese opptil ca. 1000 basepar i et strekk. Deretter kunne man i prinsippet lage et nytt startpunkt og lese 1000 nye basepar, men dette fungerte dårlig i praksis. Craig Venters lure grep var altså at han isteden klippet opp hele genomet i tilfeldige biter og laget mange kopier av disse bitene, og deretter sekvenserte han bitene hver for seg, utdyper Jakobsen.
Dermed fikk Venter i prinsippet sekvensert hvert punkt (dvs. hver base) for eksempel 50 ganger fordelt på tilfeldige biter med en lengde på ca. 1000 basepar, og bitene dekket hele den opprinnelige DNA-strengen 50 ganger. Deretter var det en forholdsvis smal sak – for en superdatamaskin som jobbet for fullt i et par uker – å legge alle bitene ved siden av hverandre og skyve dem att og fram helt til de overlappet hverandre. Slik gikk det an å regne seg fram til hvordan den opprinnelige sekvensen måtte ha sett ut - og vitenskapen som vi kjenner som genom-bioinformatikk, var født.
– Det er i prinsippet slik vi fortsatt gjør det den dag i dag. Men hastigheten har gått voldsomt oppover og kostnadene voldsomt nedover, i mange rykk og napp – styrket av diverse teknologiske gjennombrudd. Den dominerende teknologien i dag leveres fra selskapet Illumina, som vi har brukt mye her ved UiO. Men vi har også jobbet mye med maskiner fra konkurrenten PacBio. Illumina har for øvrig nettopp kjøpt PacBio, men kjøpet er ennå ikke godkjent av de amerikanske konkurransemyndighetene, forteller Jakobsen.
De tre analysemetodene
Men så var det selve lesingen, da. Professor Jakobsen forteller at den eldste teknikken gikk ut på å splitte opp den opprinnelige, doble DNA-tråden til to enkelt-tråder. Det er for øvrig akkurat det samme som foregår når cellene skal dele seg i naturen.
– I dag tar vi utgangspunkt i den ene DNA-tråden når vi bruker Illuminas metode, og sørger for at tråden lager en speilkopi av seg selv. Men ikke før vi har satt små «merkelapper» på byggesteinene som skal inngå i speilkopien! Det er egentlig snakk om fluorescens, men for enkelhets skyld kan du tenke på merkelappene som om de var farger. Vi har altså satt for eksempel en rød merkelapp på A-molekylet, en gul på C-molekylet, en blå på T-molekylet og en grønn på G-molekylet. Når den nye DNA-tråden er ferdig, går vi inn med et slags kamera som klarer å gjenkjenne de fargede merkelappene - og vips, så har vi kartlagt rekkefølgen av de fire basemolekylene.

PacBios metode gjør isteden bruk av små brikker eller chiper hvor enkeltmolekyler av DNA-strengene er plassert inne i bittesmå kamre sammen med byggesteinene til DNA. Et enzym lager en ny DNA-tråd - og så sendes det en liten laserstråle ned i hvert kammer. Denne laserstrålen bringes til å lyse på ett og ett basemolekyl av gangen. Basemolekylene har fortsatt ulike fluorescerende merkelapper og reagerer med å svinge med litt ulike frekvenser når laserstrålen tilfører dem energi. Disse ulike frekvensene kan detekteres.
Det finnes også en tredje teknologi, som nylig ble utviklet og kommersialisert av bedriften Oxford Nanopore Technologies. Kjetill S. Jakobsen holder opp et instrument på størrelse med en avlang smart-telefon, med en analyse-enhet i en brikke som er gjennomhullet av et utall små porer. De lange DNA-molekylene som skal analyseres, trekkes enkeltvis igjennom porene - som er utstyrt med bittesmå kontakter som sender ut elektriske pulser. Disse pulsene er så fintfølende at de kan kjenne igjen de ulike basemolekylenes overflatestruktur.
90-95 prosent er ganske bra
Når UiO-forskerne bruker PacBio-teknologien, pleier de å analysere hele genomet ca. 70- 80 ganger.
– Årsaken er at det alltid blir noen «hull» her og der, blant annet fordi DNA-trådene også inneholder til dels lange, repeterte sekvenser som ikke er egentlige gener. Disse sekvensene kan være så lange at vi ikke kommer gjennom dem med de teknikkene vi har i dag. Men hvis vi har klart å sette sammen 90 til 95 prosent av genomet, synes vi det er ganske bra, forteller Jakobsen.
PacBio-teknologien kan brukes til å lese opptil 100-200 ganger så lange sekvenser som Illumina-teknologien, men i det siste har også den nye nanopore-teknologien blitt en alvorlig utfordrer. Med nanoporer er det mulig å sekvensere opptil 40 000 til 60 000 baser etter hverandre, forteller Jakobsen.
Fra ti år til en uke

Mens det tok 13 år å sekvensere og kartlegge det første humane genomet, tar det i dag ca. én uke å gjennomføre dem maskinelle delen av sekvenseringen. Mer enn 10 000 mennesker var involvert i HUGO-prosjektet, mens genene hos Tore Torsk ble sekvensert av fem IBV-forskere som byttet på å være på lab'en.
Etter at den maskinelle kartleggingen av DNA-sekvensene er gjennomført, tar det i alle fall et par dager å laste de enorme datafilene ut av sekvenseringsmaskinene og over til datamaskinene som skal brukes i analysene.
– På dette stadiet sitter vi med lange strenger av de fire «bokstavene», altså A, G, C og T. Men det er nå den virkelig store jobben begynner. Først må vi løse puslespillet og sette sammen den opprinnelige DNA-strengen, ut fra de mange bitene vi har fått ut av maskinen. Og helt til slutt skal vi lete etter gener og forstå hva de lange sekvensene egentlig betyr. Det er denne delen av arbeidet som fortsatt tar tid, forteller Jakobsen.
Professor Jakobsen ser for seg at teknologien kommer til å bli enda raskere og billigere i fremtiden. Kanskje det til og med blir teknisk mulig å kartlegge DNA-sekvensen din i løpet av noen minutter, for eksempel mens du snakker med fastlegen.
– Men jeg er ikke så sikker på at fastlegene egentlig vil ha mye bruk for en slik teknologi på legekontoret. Og tolkningen av dataene vil i overskuelig framtid kreve spesiell ekspertise. Da vi begynte med DNA-kartlegging, var det jo mange som trodde at det skulle bli enkelt å finne enkeltgener som økte risikoen for enkelte sykdommer, men nå har vi isteden lært at sammenhengene er mye mer kompliserte. Blant det mangfoldet som finnes av genetisk betingede eller assosierte sykdommer er det veldig få som skyldes feil i kun ett gen, påpeker Jakobsen.
Internasjonalt samarbeid
Professor Kjetill S. Jakobsen synes uansett at DNA-sekvensering er noe av det morsomste og viktigste som finnes. Nå jobber han blant annet for at Norge og CEES skal bli med som en viktig aktør i det store, internasjonale prosjektet Earth Biogenome Project (EBP). Målsettingen er intet mindre enn å sekvensere, katalogisere og karakterisere genomene hos alle verdens eukaryote arter (organismer med cellekjerne) i løpet av de kommende ti årene.
– Det blir både et stort og viktig prosjekt, og jeg har stor tro på internasjonalt samarbeid. Men av og til kan slikt samarbeid gi opphav til noen pussige episoder, forteller Jakobsen.
DNA blir, som tidligere nevnt, veldig godt konservert i etanol. For noen år siden skulle UiO-forskerne undersøke genene hos en russisk gaupestamme og sendte små prøveglass med 96 prosent etanol til de russiske samarbeidspartnere. Etter en stund kom prøveglassene tilbake med små vevsprøver fra gaupene. Så langt, alt vel.
– Men de russiske forskerne hadde drukket opp etanolen i prøveglassene og fylt dem opp med vann! Vann konserverer ikke DNA i det hele tatt, så de gaupe-prøvene kunne vi ikke bruke. Da begynte vi å sende prøveglass med isopropanol (en alkohol som blant annet brukes i frostvæske) istedenfor etanol til Russland, og vi la med et følgebrev hvor det sto uttrykkelig at dette må dere ikke bruke i kaffen! Etter det gikk samarbeidet på skinner, ler Jakobsen.
Kontaktperson:
Professor Kjetill S. Jakobsen, Institutt for biovitenskap og CEES
Mer informasjon:
Trine Nickelsen: Løste DNA-gåten – men falt ut av historien (Apollon, 12. mars 2013)
Naturfagsenteret: Isoler ditt eget DNA





